合作客户/
拜耳公司 |
同济大学 |
联合大学 |
美国保洁 |
美国强生 |
瑞士罗氏 |
相关新闻Info
推荐新闻Info
-
> 长庆油田陇东地区的CQZP-1助排剂表/界面张力测量及现场应用(三)
> 长庆油田陇东地区的CQZP-1助排剂表/界面张力测量及现场应用(二)
> 长庆油田陇东地区的CQZP-1助排剂表/界面张力测量及现场应用(一)
> 液膜断裂点与电压最大值在表面张力测量中的对比研究(二)
> 液膜断裂点与电压最大值在表面张力测量中的对比研究(一)
> 表面张力与表面张力系数测量:概念、方法与科学意义
> 微重力下二极对非均匀旋转磁场控制半浮区液桥表面张力对流的数值研究(下)
> 微重力下二极对非均匀旋转磁场控制半浮区液桥表面张力对流的数值研究(上)
> 胆汁酸通过改变肺泡表面张力调节呼吸功能的机制研究——讨论、结论、展望
> 胆汁酸通过改变肺泡表面张力调节呼吸功能的机制研究——结果
Delta-8使用新方法测试CMC,而不是表面张力测试法——讨论、致谢!
来源:Unisense 浏览 2523 次 发布时间:2021-09-03
讨论
在这里,我们提出了一种新颖的、高通量的、基于非细胞的使用荧光法预测PLD的体外筛选方法通过监测磷脂的CMC进行探测,有无与测试物品的交互。而朗缪尔平衡方法27提供了通过CMC评估PLD的可靠途径转变,当前的荧光方法提供了一种独特的替代方法本机用于预测PLD风险。这种与微孔板兼容的平台形式更具成本效益,更易于操作,具有更高的通量,可修改用于早期药物的PLD风险评估发现。所选的荧光探针Prodan适用于脂质和测试的CMC测定形成胶束的化合物。我们清楚地证明了体内与体外或基于非细胞的对比与细胞检测相关性使用当前方法,总体一致性为91%当应用合适的截止值(CMCDL/CMCL=0.75 in本试验)。与两个in silico模型相比,这更好我们也应用于我们的验证集,其一致性Ploemen19和Tomizawa20模型的值分别为76%和80%,分别。敏感性(PLD诱导物鉴定为阳性)和选择性(非PLD诱导剂鉴定为阴性)模型分别为92%和90%,也优于两种计算机模型(表3)。硅片和体外分析利用了测试的固有物理化学特性使它们对磷脂更亲和的化合物pids;然而,体外模型也可以确定药物浓度对药物脂质相互作用的影响可通过计算机模型进行预测。此外,空间化学分子和电荷的空间分布及其极性和非极性部分可以在反应性中发挥作用测试化合物和这些因素没有被硅胶模型。此外,这个体外模型正确分配庆大霉素和红霉素等非CAD药物的PLD诱导潜力,因为体外模型评估了总体因此,测试化合物和脂质之间的相互作用是比硅模型更先进。反之,对于化合物在所需测试浓度下存在溶解度问题,另一种选择应确定溶剂,否则数据可能会产生误导。在硅胶模型没有这个障碍,可以应用于任何具有明确结构的化合物。
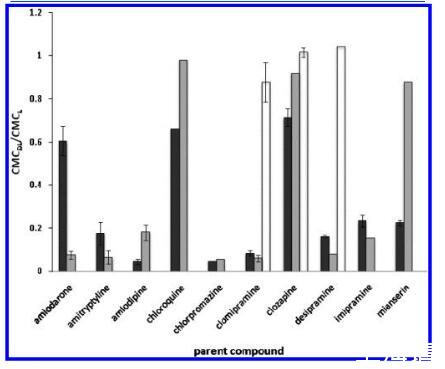
图5.CMC偏移(由CMC之间的比率表示)药物-脂质复合物(DL)和单独脂质的CMC(L):CMCDL/CMCL)的测试化合物(黑色)及其主要代谢物(灰色、初级代谢物;白色,次生代谢物)在pH 7.2中分析HEPES缓冲液。使用的药物浓度为1 mM,除非另有说明见表2。误差线被分配给那些n g3测试的化合物。对于氯氮平和米安色林,均为0.91和0.091 mM用于测试,但仅来自1 mM测试的数据被策划。
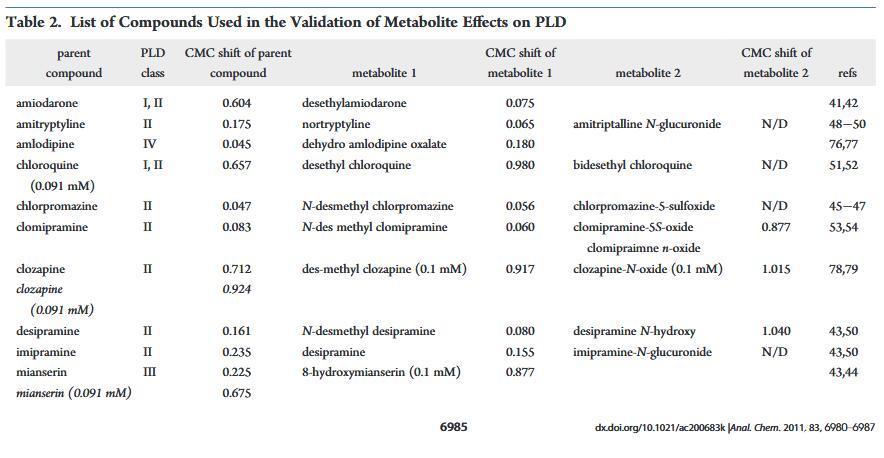
表2
据报道,药代动力学对PLD有影响,因为剂量、24、33、36代谢、32、37?39和处置10该药物均影响体内PLD的严重程度。创建一个成功的候选人需要清楚地了解PLD的原因,从而重新设计化学结构或评估合适的治疗窗。
PLD所需的最低剂量不同于磅到复合,有时取决于性别。24将体内剂量与体外试验浓度直接挂钩可能具有挑战性,因为药物的血浆浓度可能受其渗透性、溶解性、代谢、所用赋形剂、给药途径、用药频率摄入量等等。但通过监测浓度测试分子对PLD能力的依赖性,即不太可能是线性的,如图4所示,并且很难估计,我们可以确定之间的潜在关系剂量和脂质结合,从而评估安全窗口体内试验数量有限。
已经有许多出版物描述了PLD药物代谢产物所致。其中,初级代谢轻的胺碘酮,去乙基胺碘酮,已经讨论过14,38-40优先积累代谢物已观察到胺碘酮过量,尤其是短期39这为我们的检测数据提供了可能的解释其中去乙基胺碘酮是胺碘酮的主要代谢物,41,42表现出更高的PLD诱导潜力。相似地,8-羟基米安色林之间的CMC位移差异,米安色林、43,44和米安色林的主要氧化代谢物比其他两个之间的差异更显着成对的亲本和代谢物。由于溶解度的限制,父母和代谢组的剂量浓度不同氯氮平和米安色林。这可能是导致观察到的CMC偏移的显着差异母体和代谢物之间。在较低浓度下测试时相比之下,氯氮平从低电位诱导剂转变为非诱导剂,而米安色林仍然作为诱导剂但具有更高的CMCDL/CMCL值。值得注意的是,目前测试集中的大多数CAD被报告为氧化代谢其相应的脱烷基初级代谢物。为了了解相应初级的PLD电位代谢物,我们测试了额外的代谢物。之间测试的代谢物是初级去甲基代谢物氯丙嗪、45?47阿米替林、48?50丙咪嗪、43,50和pri-氯喹的玛丽和次级脱乙基代谢物。51,52此外,由杂原子氧化形成的氧化代谢物地昔帕明43,50和氯胺酮的重释和脱烷基代谢物pramine53,54进行了评估。与其他CAD一样,代谢物可以如果它们具有合适的理化性质,则诱导PLD。32相反,代谢物将失去结合能力如果CAD特征丢失,则使用脂质,例如用脂质观察氯米帕明N-氧化物和地昔帕明N-羟基(图4和表2)。
表3

众所周知,流通的水平和时间可能不同的代谢物以及从一种药物到另一种药物的变化;然而,循环的量(以摩尔计)或剂量当量代谢物可能永远不会大于给药的代谢物特雷德药。在本研究中,代谢物在与他们的父母相比,相同或更低的浓度基于它们的溶解度。应该注意的是该检测是作为一种前瞻性筛选工具而开发的;那里-因此,基本前提是测试代谢物在与母体浓度相同。在以下情况下代谢物的PLD潜力值得关注,浓度可以与相关元进行相关研究博莱特。然而,这样的研究超出了该研究的范围。目前的调查。层状体的识别通过透射电子显微镜(TEM)是第一个适应症溶酶体参与PLD9并表明溶酶体pH值可能是一个关键因素;20然而,最初没有直接溶酶体受累的起源和功能的证据。3 A Max field和MacGraw进行的研究表明在存在下形成的内体的快速隔离变成溶酶体的磷脂生成药物;55然而,它溶酶体的酸性pH值可能会改变脂质和目标药物之间相互作用的严重程度溶酶体包涵体的形成。由于这些原因,我们相信研究这两种pH条件是有价值的,因此获得更全面的风险评估。在我们的验证集中,大多数化合物在pH 7.4和4.8除了米安色林、胺碘酮和华法林,以及所有这三种通过不同的体内和实验证明化合物是PLD诱导剂细胞检测。
该测定显示出高重现性和可预测性。类似于其他无法替代体内的体外测定确认结果的测量,这更便宜,更少耗时的荧光方法可以提供有用的有关药物与脂质相互作用的早期信息。此外,这种方法可用于对候选人进行排名,协助药物设计,并估计潜在风险。
AUTHOR INFORMATION
Corresponding Author
*Phone:617-871-7143.E-mail:LipingX.Zhou Novartis.com.
Present Addresses
§Department of Chemistry,University of Colorado-Denver,Denver,CO 80205.
ACKNOWLEDGMENT
We would like to thank Dr.Pavel Landsman for his insight onfluorescent dye probe selections,design of the experiments,and helpful discussions.Our thanks also go to Mr.Alan Bushey from Novartis IT for development of the data processing Microsoft Excel worksheet.
REFERENCES
Nelson,A.A.;Fitzhugh,O.G.Arch.Pathol.1948,45(4),454–462.
Hostetler,K.Y.;Reasor,M.;Yazaki,P.J.J.Biol.Chem.1985,260(1),215–219.
Kodavanti,U.P.;Mehendale,H.M.Pharmacol.Rev.1990,42327–354.
Vanhaelst,U.J.G.M.;Elving,L.;Hoefnagels,W.Ultramicro-scopy 1986,19(1),107–108.
Schneider,P.Arch.Toxicol.1992,66(1),23–33.
Halliwell,W.H.Toxicol.Pathol.1997,25(1),53–60.
Reasor,M.J.;Hastings,K.L.;Ulrich,R.G.Expert Opin.Drug Saf.
2006,5(4),567–583.
Martin,W.J.;Kachel,D.L.;Vilen,T.;Natarajan,V.J.Pharmacol.Exp.Ther.1989,251(1),272–278.
(9)Anderson,N.;Borlak,J.FEBS Lett.2006,580(23),5533–5540.
Hanumegowda,U.M.;Wenke,G.;Regueiro-Ren,A.;Yorda-nova,R.;Corradi,J.P.;Adams,S.P.Chem.Res.Toxicol.2010,23(4),749–755.
Reasor,M.J.;Castranova,V.Exp.Mol.Pathol.1981,35(3),359–369.
Reasor,M.J.;Walker,E.R.Exp.Mol.Pathol.1981,35(3),370–379.
Pelletier,D.J.;Gehlhaar,D.;Tilloy-Ellul,A.;Johnson,T.O.;Greene,N.J.Chem.Inf.Model.2007,47(3),1196–1205.
Sun,E.L.;Petrella,D.K.;McCloud,C.M.;Cramer,C.T.;Reasor,M.J.;Ulrich,R.G.In Vitro Toxicol.1997,10(4),459–470.
Watanabe,Y.;Watanabe,K.;Tashiro,I.;Enomoto,Y.
J.Electron Microsc.1971,20(3),255.
Tashiro,Y.;Watanabe,Y.;Enomoto,Y.Acta Pathol.Jpn.1983,33(5),929–942.
Monteith,D.K.;Morgan,R.E.;Halstead,B.Expert Opin.Drug Metab.Toxicol.2006,2(5),687–696.
Nonoyama,T.;Fukuda,R.Toxicol.Pathol.2008,21(1),9–24.
Ploemen,J.P.H.T.;Kelder,J.;Hafmans,T.;Van De Sandt,H.;van Burgsteden,J.A.;Salemink,P.J.M.;Van Esch,E.Exp.Toxicol.Pathol.2004,55(5),347–355.
Tomizawa,K.;Sugano,K.;Yamada,H.;Horii,I.J.Toxicol.Sci.
2006,31(4),315–324.
Laurent,G.;Carlier,M.B.;Rollman,B.;Vanhoof,F.;Tulkens,P.Biochem.Pharmacol.1982,31(23),3861–3870.
Nioi,P.;Perry,B.K.;Wang,E.J.;Gu,Y.Z.;Snyder,R.D.
Toxicol.Sci.2007,99(1),162–173.
Morelli,J.K.;Buehrle,M.;Pognan,F.;Barone,L.R.;Fieles,W.;Ciaccio,P.J.Cell Biol.Toxicol.2006,22(1),15–27.
Miyamoto,S.;Matsumoto,A.;Mori,I.;Horinouchi,A.Toxicol.Mech.Methods 2009,19(8),477–485.
Natalie,M.;Margino,S.;Erik,H.;Annelieke,P.;Geert,V.;Philippe,V.Toxicol.in Vitro 2009,23(2),217–226.
Sawada,H.;Taniguchi,K.;Takami,K.Toxicol.in Vitro 2006,20
(8),1506–1513.
Vitovic,P.;Alakoskela,J.M.;Kinnunen,P.K.J.J.Med.Chem.
2008,51(6),1842–1848.
De Vendittis,E.;Palumbo,G.;Parlato,G.;Bocchini,V.Anal.Biochem.1981,115,278–286.
Deb,N.;Shannigrahi,M.;Bagchi,S.J.Phys.Chem.B 2008,112
2868–2873.
Moyano,F.;Silber,J.J.;Correa,N.M.J.Colloid Interface Sci.
2008,317,332–345.
Moyano,F.;Biasutti,M.A.;Silber,J.J.;Correa,N.M.J.Phys.Chem.B 2006,110,11838–11846.
Gum,R.J.;Hickman,D.;Fagerland,J.A.;Heindel,M.A.;Gagne,G.D.;Schmidt,J.M.;Michaelides,M.R.;Davidsen,S.K.;Ulrich,R.G.Biochem.Pharmacol.2001,62(12),1661–1673.
Wilson,B.D.;Clarkson,C.E.;Lippmann,M.N.Am.Rev.Respir.Dis.1991,143,1110–1114.
Lahoti,S.;Lee,W.M.Gastroenterol.Clin.North Am.1995,24
(4),907–922.
Kita,N.;Sugihara,N.;Furuno,K.J.Pharmacobiodyn.1992,15
(4),181–189.
Giuliano,R.A.;Paulus,G.J.;Verpooten,G.A.;Pattijn,V.M.;Pollet,D.E.;Nouwen,E.J.;Carlier,M.B.;Maldague,P.;Tulkens,P.M.;Debroe,M.E.Hum.Toxicol.1984,3(5),432.
Kacew,S.;Narbaitz,R.;Ruddick,J.A.;Villeneuve,D.C.Exp.Mol.Pathol.1981,35(1),98–107.
Kodavanti,U.P.;Mehendale,H.M.Am.J.Respir.Cell Mol.Biol.
1991,4(4),369–378.
Antonini,J.M.;Reasor,M.J.Biochem.Pharmacol.1991,42
S151–S156.
Kannan,R.;Sarma,J.S.M.;Guha,M.;Venkataraman,K.Eur.J.Pharmacol.1990,183(6),2452–2453.
Nattel,S.J.Cardiovasc.Pharmacol.1986,8(4),771–777.
Latini,R.;Tognoni,G.;Kates,R.E.Clin.Pharmacokinet.1984,9(2),136–156.
Riley,R.J.;Roberts,P.;Kitteringham,N.R.;Park,B.K.
Biochem.Pharmacol.1990,39(12),1951–1958.
Lambert,C.;Park,B.K.;Kitteringham,N.R.Biochem.Pharmacol.1989,38(17),2853–2858.
Wojcikowski,J.;Boksa,J.;Daniel,W.A.Biochem.Pharmacol.
2010,80(8),1252–1259.
Stevens,J.C.;Shipley,L.A.;Cashman,J.R.;Vandenbranden,M.;Wrighton,S.A.Drug Metab.Dispos.1993,21(5),753–760.
Hartmann,F.;Gruenke,L.D.;Craig,J.C.;Bissell,D.M.Drug Metab.Dispos.1983,11(3),244–248.
Gupta,R.N.;Molnar,G.;Hill,R.E.;Gupta,M.L.Clin.Biochem.1976,9(5),247–251.
Prox,R.;Breyer-Pfaff,U.Drug Metab.Dispos.1987,15(6),890–896.
Vandemark,F.L.;Adams,R.F.;Schmidt,G.J.Clin.Chem.
1978,24(1),87–91.
Kim,K.A.;Park,J.Y.;Lee,J.S.;Lim,S.Arch.Pharm.Res.2003,26(8),631–637.
Projean,D.;Baune,B.;Farinotti,R.;Flinois,J.P.;Beaune,P.;Taburet,A.M.;Ducharme,J.Drug Metab.Dispos.2003,31(6),748–754.
Wu,Z.L.;Huang,S.L.;Ou-Yang,D.S.;Xu,Z.H.;Xie,H.G.;Zhou,H.H.Zhongguo Yao Li Xue Bao 1998,19(5),433–436.
Nielsen,K.K.;Flinois,J.P.;Beaune,P.;Brosen,K.J.Pharmacol.Exp.Ther.1996,277(3),1659–1664.
Maxfield,F.;McGraw,T.Nat.Rev.Mol.Cell Biol.2004,5
121–132.
Sawada,H.;Takami,K.;Asahi,S.Toxicol.Sci.2005,83(2),282–292.
Toubeau,G.;Nonclercq,D.;Zanen,J.;Lambricht,P.;Tulkens,
P.M.;Heuson Stiennon,J.A.;Laurent,G.Kidney Int.1991,40(4),691–699.
Poucell,S.;Valencia,P.;Ireton,J.;Downar,E.;Patterson,J.;Blendis,L.;Phillips,M.J.Hepatology 1983,3(5),805.
Poucell,S.;Ireton,J.;Valenciamayoral,P.;Downar,E.;Larratt,L.;Patterson,J.;Blendis,L.;Phillips,M.J.Gastroenterology 1984,86(5),926–936.
Goldman,I.S.;Winkler,M.L.;Raper,S.E.;Barker,M.E.;Keung,E.;Goldberg,H.I.;Boyer,T.D.Am.J.Roentgenol.1985,144(3),541–546.
Riva,E.;Marchi,S.;Pesenti,A.;Bizzi,A.;Cini,M.;Veneroni,E.;Tavbani,E.;Boeri,R.;Bertani,T.;Latini,R.Biochem.Pharmacol.1987,36(19),3209–3214.
Hruban,Z.Environ.Health Perspect.1984,55,53–76.
Kodavanti,U.P.;Lockard,V.G.;Mehendale,H.M.J.Biochem.Toxicol.1990,5(4),245–251.
Lullmann-Rauch,R.;Nassberger,L.Acta Pharmacol.Toxicol.
1983,52,161–167.
Yano,T.;Ohmori,S.;Igarashi,T.;Ueno,K.;Kitagawa,H.Jpn.J.Pharmacol.1987,43,207.
Gray,J.E.;Weaver,R.N.;Stern,K.F.;Phillips,W.A.Toxicol.Appl.Pharmacol.1978,45(3),701–711.
Lullmann-Rauch,R.;Reil,G.H.Arch.Pharmacol.1974,
285,175–184.
(68)Valodia,P.N.;Syce,J.A.Med.Sci.Res.1998,26(7),491–493.
Gonzalezrothi,R.J.;Zander,D.S.;Ros,P.R.Chest 1995,107
(6),1763–1765.
Auberttulkens,G.;Vanhoof,F.Arch.Int.Physiol.Biochim.Biophys.1978,86(2),403–405.
Pakuts,A.P.;Parks,R.J.;Paul,C.J.;Bujaki,S.J.;Mueller,R.W.
Res.Commun.Chem.Pathol.Pharmacol.1990,67(1),55–62.
Staubli,W.;Schweizer,W.;Suter,J.Exp.Mol.Pathol.1978,
28,177–195.
Glaumann,H.;Bronner,U.;Ericsson,O.;Gustaffson,L.L.;Rombo,L.Pharmacol.Toxicol.1994,74,17–22.
Lullmann,H.;Lullmann-Rauch,R.Klin.Wochenschr.1978,
56,309–310.
Kasahara,T.;Tomita,K.;Murano,H.;Harada,T.;Tsubakimoto,K.;Ogihara,T.;Ohnishi,S.;Kakinuma,C.Toxicol.Sci.2006,90(1),133–141.
Beresford,A.P.;McGibney,D.;Humphrey,M.J.;Macrae,P.V.;Stopher,D.A.Xenobiotica 1988,18(2),245–254.
Stopher,D.A.;Beresford,A.P.;Macrae,P.V.;Humphrey,M.J.
J.Cardiovasc.Pharmacol.1988,12,S55–S59.
Pirmohamed,M.;Williams,D.;Madden,S.;Templeton,E.;Park,B.K.J.Pharmacol.Exp.Ther.1995,272(3),984–990.
Tugnait,M.;Hawes,E.M.;McKay,G.;Eichelbaum,M.;Midha,K.K.Chem.Biol.Interact.1999,118(2),171–189.
Delta-8使用新方法测试CMC,而不是表面张力测试法——摘要
Delta-8使用新方法测试CMC,而不是表面张力测试法——方法










